紧接上篇基础教程,本文将继续深入,以蛋白质数据库 (PDB) 中的 1HEW 复合物结构为例,演示 PyMOL 中对象列表面板的进阶操作。
4. 对象列表面板功能介绍
对象列表面板是 PyMOL 中进行可视化控制和交互的核心区域,熟练掌握其功能将极大提升你的分析效率。
1. 理解面板构成
在面板中,你通常会看到三个主要项目:“All”、当前加载的蛋白质名称(此处为“1hew”)以及“(sele)”。其中,“(sele)” 是 selection 的缩写,代表当前鼠标正在选择或高亮显示的对象。
请注意,面板左上角的下拉菜单允许你更改选择对象的粒度,可以是原子 (atoms)、残基 (residues)、链 (chains) 或整个分子 (molecules)。你所选择的氨基酸残基会同步在下方显示的蛋白质序列中被高亮。如果需要选择一段连续的残基,只需在序列窗口中点击并拖拽,或者逐个点击目标残基即可。
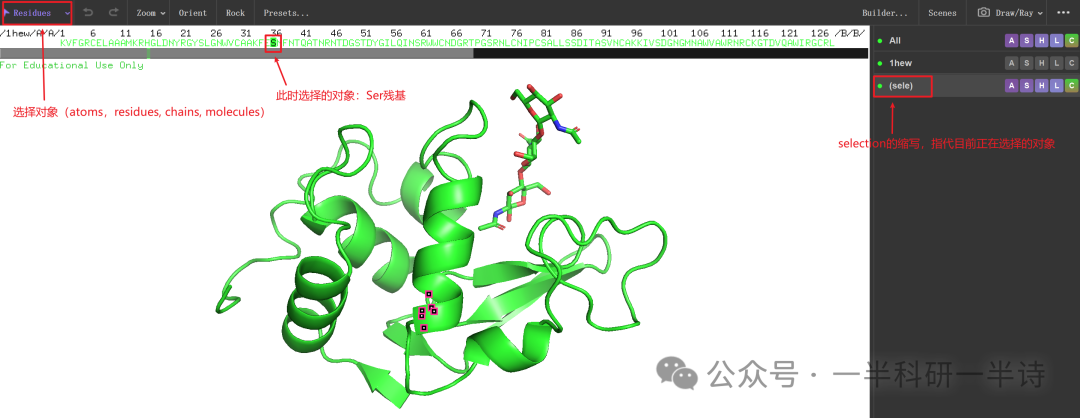
2. 核心操作按钮:A, S, H, L, C
每个对象名称的右侧都有一排字母按钮:A, S, H, L, C。它们是实现对蛋白质复合物进行各种基础操作的快捷入口。
| 按钮 |
全称 |
主要功能 |
备注 |
| A |
Action |
对当前对象进行整体操作,如调整视角、显示相互作用、结构对齐、删除/复制/重命名对象、氢原子相关操作等。 |
- |
| S |
Show |
以不同样式展示蛋白质、侧链、主链、配体等。 |
注意区分:show+style 是在现有样式上叠加新样式;show as+style 会清除原有样式,仅显示新样式。 |
| H |
Hide |
隐藏由 Show 展示的各种样式、主链、侧链、水分子或氢原子等。 |
hide everything 会隐藏包括标签在内的一切。 |
| L |
Label |
标记感兴趣的特定氨基酸残基、原子等,用于在3D视图中直接显示其名称或编号。 |
- |
| C |
Color |
为对象着色,可以按原子、链、二级结构、B因子等多种属性进行颜色分配。 |
- |
3. 操作的作用范围
这是一个关键概念:对“1hew”或“(sele)”等特定对象进行的 A, S, H, L, C 操作,仅作用于该对象本身。而对顶部的“All”进行相同的操作,则会同时作用于对象列表中的所有对象(例如“1hew”和水分子等),实现批量控制。
4. 显示与隐藏控制
对象列表左侧的圆点用于控制该对象在3D视图中的可见性。绿点表示显示,灰点表示隐藏。通过点击圆点,你可以快速开关某个特定对象(如某条蛋白链或配体)的显示,便于聚焦分析。
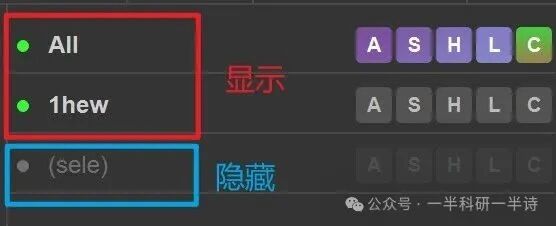
5. 综合练习
现在,让我们运用以上学到的功能,完成一个简单的综合练习,目标是将蛋白质1HEW的R61和N106两个残基的侧链以特定样式突出显示。
操作步骤如下,你可以参考下方的示意图同步进行:
File > Get PDB > 1HEW # 从PDB数据库获取蛋白结构Display > Background > White # 将背景设置为白色Display > Sequence (或点击 SEQ 按钮) # 显示蛋白质序列窗口- 在对象列表中,对
1hew 点击 A > remove waters # 移除结构中的水分子
- 在序列窗口中,选择目标残基(如R61和N106),此时
(sele) 会代表你的选择。
- 对
(sele) 点击 S > side chain > sticks # 将选中残基的侧链显示为棍状模型
- 对
(sele) 点击 L > residues (one letter) # 为选中的残基添加单字母标记
- 对
(sele) 点击 C > by element > 选择紫色 C 骨架 # 将选中残基的侧链碳骨架改为紫色

通过这个练习,你应该能更直观地理解对象列表面板中各个按钮的协同工作方式。掌握这些基础操作后,你就能在 云栈社区 的技术文档板块中,更高效地探索和学习更复杂的PyMOL分析技巧与脚本编写了。 |