WebLogo 是生物信息学领域一款经典的工具,专门用于将 DNA、RNA 或蛋白质的多序列比对结果进行可视化,直观展示每个位点上碱基或氨基酸的保守性。它通过堆叠字母的大小和颜色来反映不同残基的出现频率,是研究序列功能与结构的关键辅助手段。本文将以同源蛋白质为例,手把手教你如何从序列比对开始,生成一张专业的序列保守性 Logo 图。
1. 蛋白的多序列比对
在 FigTree 系统发育树分析教程(一) 中,我们曾介绍过使用 MUSCLE 进行同源蛋白序列的比对。WebLogo 支持上传 .aln(ClustalW格式)或 .fa(FASTA格式)的比对结果文件,因此我们只需在 MUSCLE 的输出步骤中选择其中一种格式并下载即可。

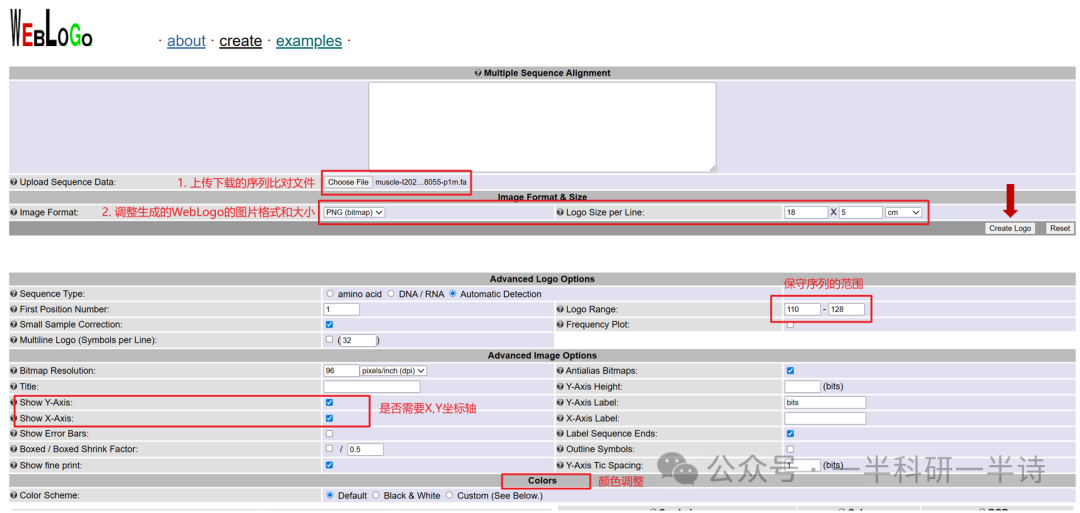
2. WebLogo的生成与参数设置
WebLogo 的在线界面非常简洁。操作核心分三步:首先上传刚才下载的序列比对文件;接着调整生成图片的格式、尺寸等基本参数;最后,根据你关注的保守区域,调整序列范围、坐标轴显示以及颜色方案等高级设置。
这一切数据处理和可视化逻辑,都离不开底层计算能力的支撑。在 智能 & 数据 & 云 领域,高效的计算资源是处理海量生物序列、运行复杂比对算法的基石。
生成多个保守区域的 Logo 图时,可以分别生成,然后在 PowerPoint 或 Adobe Illustrator 等软件中进行排版和美化。
3. 结果解读
下图展示的是一个蛋白质序列片段(第110至128位氨基酸)的 WebLogo 生成结果。纵坐标(bits)表示信息熵,数值越高,代表该位点的保守性越强。每个位置堆叠的字母代表该位点可能出现的氨基酸,字母的大小直观反映了其出现的相对频率。

从图中我们可以清晰地识别出高度保守的位点。这类精细的序列分析是理解蛋白质功能、进行理性设计的关键一步,也体现了现代 人工智能 与数据科学在生命科学领域的深度交叉应用。
希望这篇基于实操的指南能帮助你快速上手 WebLogo。如果你想了解更多生物信息学或数据可视化相关的工具技巧,欢迎来 云栈社区 交流探讨。 |