本文深入探讨UNet网络架构及其核心变体,涵盖理论原理、数学推导、PyTorch代码实现与实验对比,面向具有深度学习基础的专业读者。
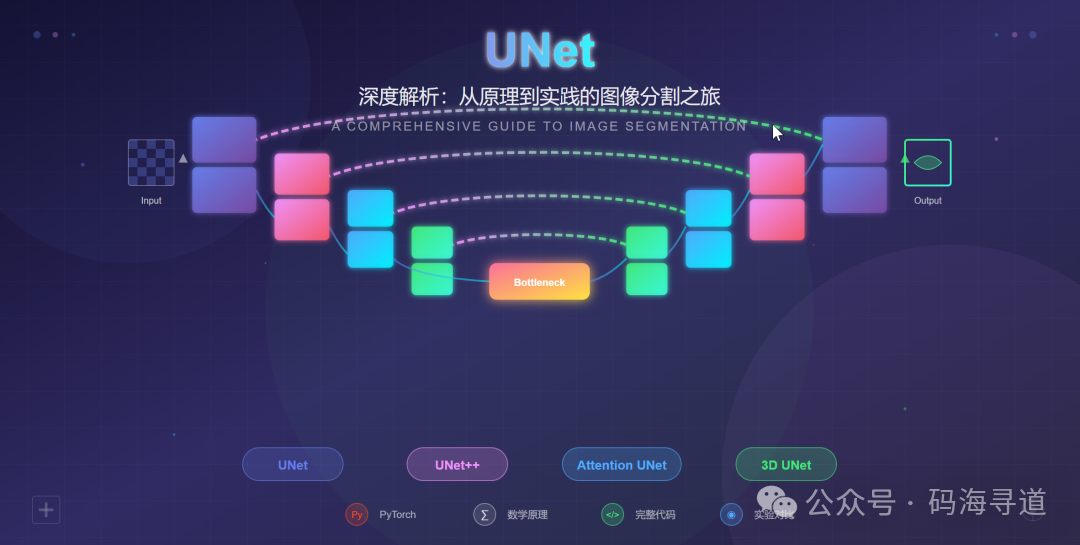
目录
- 引言
- UNet原理详解
- UNet++ (Nested UNet)
- Attention UNet
- 3D UNet
- 实验对比
- 应用场景
- 总结与展望
- 参考文献
1. 引言
1.1 图像分割任务背景
图像分割是计算机视觉领域的核心任务之一,其目标是将图像划分为多个具有语义意义的区域,为每个像素分配类别标签。根据任务粒度的不同,图像分割可分为:
- 语义分割:为每个像素分配类别标签,不区分同类实例。
- 实例分割:区分同类别的不同实例。
- 全景分割:结合语义分割与实例分割。
在医学影像、自动驾驶、遥感分析等领域,精确的图像分割是后续分析与决策的基础。
1.2 UNet的历史意义
2015年,Olaf Ronneberger等人在MICCAI会议上发表了具有里程碑意义的论文《U-Net: Convolutional Networks for Biomedical Image Segmentation》。该论文提出的UNet架构,凭借其优雅的编码器-解码器对称结构和创新的跳跃连接机制,在生物医学图像分割任务中取得了突破性成果。UNet已成为图像分割领域最具影响力的基础架构之一,催生了众多变体网络。
1.3 本文内容概览
本文将系统性地介绍UNet及其核心变体,包括基础UNet、通过密集跳跃连接优化特征融合的UNet++、引入注意力机制的Attention UNet以及扩展至三维体积数据处理的3D UNet。每个架构都将从设计动机、网络结构、数学原理、代码实现等多个维度进行深入剖析。
2. UNet原理详解
2.1 网络架构
UNet的架构呈现标志性的U形结构,由编码器(收缩路径)、解码器(扩展路径)和跳跃连接三部分组成。
2.1.1 编码器(Contracting Path)
编码器负责提取图像的多尺度特征,采用典型的卷积神经网络结构:
- 双卷积块:每层包含两个3×3卷积(无填充),后接ReLU激活。
- 下采样:2×2最大池化,步长为2。
- 通道倍增:每次下采样后,特征通道数翻倍(64→128→256→512→1024)。
2.1.2 解码器(Expanding Path)
解码器负责将低分辨率特征图逐步恢复到原始分辨率:
- 上采样:2×2转置卷积(或双线性插值 + 1×1卷积)。
- 特征拼接:将编码器对应层的特征图与上采样结果在通道维度拼接。
- 双卷积块:两个3×3卷积处理拼接后的特征。
2.1.3 瓶颈层(Bottleneck)
位于U形结构底部,是编码器与解码器的连接点,包含两个3×3卷积层,特征通道数达到最大值(1024)。
2.2 核心创新:跳跃连接
跳跃连接是UNet最重要的设计创新,它直接将编码器的特征图传递给解码器对应层。
2.2.1 设计动机
在传统的编码器-解码器架构中,经过多次下采样后,空间细节信息会严重丢失。跳跃连接通过保留低层的边缘、纹理等细节信息,并与高层的语义特征进行融合,有效解决了空间信息丢失和语义鸿沟问题,同时提供了额外的梯度路径,有助于缓解梯度消失。
2.2.2 特征融合机制
UNet采用通道拼接(Concatenation)方式融合特征:x_decoder = concat(up(x_deeper), x_encoder)。与ResNet的逐元素相加不同,通道拼接保留了两个特征图的完整信息,给予网络更大的学习自由度。
2.3 损失函数
2.3.1 交叉熵损失(Cross-Entropy Loss)
像素级分类的标准损失函数,适用于各类别样本均衡的场景。
2.3.2 Dice Loss
针对医学影像分割中常见的类别不平衡问题(如小目标肿瘤)设计,基于Dice系数。
2.3.3 组合损失
实践中常采用组合损失策略,例如:Loss = α * BCE_Loss + β * Dice_Loss,以兼顾整体像素分类精度和特定区域的重叠度。
2.4 PyTorch代码实现
以下是UNet的模块化PyTorch实现:
import torch
import torch.nn as nn
import torch.nn.functional as F
class DoubleConv(nn.Module):
"""双卷积块:(Conv2d -> BN -> ReLU) × 2"""
def __init__(self, in_channels, out_channels, mid_channels=None):
super().__init__()
if mid_channels is None:
mid_channels = out_channels
self.double_conv = nn.Sequential(
nn.Conv2d(in_channels, mid_channels, kernel_size=3, padding=1, bias=False),
nn.BatchNorm2d(mid_channels),
nn.ReLU(inplace=True),
nn.Conv2d(mid_channels, out_channels, kernel_size=3, padding=1, bias=False),
nn.BatchNorm2d(out_channels),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.double_conv(x)
class Down(nn.Module):
"""下采样模块:MaxPool -> DoubleConv"""
def __init__(self, in_channels, out_channels):
super().__init__()
self.maxpool_conv = nn.Sequential(
nn.MaxPool2d(2),
DoubleConv(in_channels, out_channels)
)
def forward(self, x):
return self.maxpool_conv(x)
class Up(nn.Module):
"""上采样模块:UpConv -> Concat -> DoubleConv"""
def __init__(self, in_channels, out_channels, bilinear=True):
super().__init__()
if bilinear:
self.up = nn.Upsample(scale_factor=2, mode='bilinear', align_corners=True)
self.conv = DoubleConv(in_channels, out_channels, in_channels // 2)
else:
self.up = nn.ConvTranspose2d(in_channels, in_channels // 2, kernel_size=2, stride=2)
self.conv = DoubleConv(in_channels, out_channels)
def forward(self, x1, x2):
"""
x1: 来自解码器上一层的特征图
x2: 来自编码器对应层的特征图(跳跃连接)
"""
x1 = self.up(x1)
# 处理输入尺寸不匹配的情况
diff_y = x2.size()[2] - x1.size()[2]
diff_x = x2.size()[3] - x1.size()[3]
x1 = F.pad(x1, [diff_x // 2, diff_x - diff_x // 2,
diff_y // 2, diff_y - diff_y // 2])
# 通道拼接
x = torch.cat([x2, x1], dim=1)
return self.conv(x)
class OutConv(nn.Module):
"""输出卷积层"""
def __init__(self, in_channels, out_channels):
super().__init__()
self.conv = nn.Conv2d(in_channels, out_channels, kernel_size=1)
def forward(self, x):
return self.conv(x)
class UNet(nn.Module):
"""
UNet 完整实现
Args:
n_channels: 输入图像通道数
n_classes: 输出分割类别数
bilinear: 是否使用双线性插值上采样
"""
def __init__(self, n_channels, n_classes, bilinear=False):
super().__init__()
self.n_channels = n_channels
self.n_classes = n_classes
self.bilinear = bilinear
# 编码器
self.inc = DoubleConv(n_channels, 64)
self.down1 = Down(64, 128)
self.down2 = Down(128, 256)
self.down3 = Down(256, 512)
factor = 2 if bilinear else 1
self.down4 = Down(512, 1024 // factor)
# 解码器
self.up1 = Up(1024, 512 // factor, bilinear)
self.up2 = Up(512, 256 // factor, bilinear)
self.up3 = Up(256, 128 // factor, bilinear)
self.up4 = Up(128, 64, bilinear)
self.outc = OutConv(64, n_classes)
def forward(self, x):
# 编码器路径
x1 = self.inc(x) # 64 channels
x2 = self.down1(x1) # 128 channels
x3 = self.down2(x2) # 256 channels
x4 = self.down3(x3) # 512 channels
x5 = self.down4(x4) # 1024 channels (瓶颈层)
# 解码器路径(带跳跃连接)
x = self.up1(x5, x4) # 512 channels
x = self.up2(x, x3) # 256 channels
x = self.up3(x, x2) # 128 channels
x = self.up4(x, x1) # 64 channels
logits = self.outc(x)
return logits
(代码较长,此处继续展示损失函数及使用示例,DiceLoss和CombinedLoss类定义略)
3. UNet++ (Nested UNet)
3.1 设计动机
尽管UNet表现出色,但其跳跃连接存在语义鸿沟问题:编码器浅层特征(边缘、纹理)与深层特征(高级语义)差异巨大,直接拼接可能导致特征融合效率低下和边界定位精度受限。
3.2 网络架构
UNet++ 的核心创新是引入密集跳跃连接和深度监督机制。它通过一组嵌套的、密集的跳跃连接路径,让解码器节点能够聚合来自多个编码器层的、经过不同程度解码的特征,从而渐进式地弥合语义鸿沟。
3.3 PyTorch代码实现
(此处展示UNet++的核心结构定义,ConvBlock类及UNetPlusPlus类的__init__和forward方法关键部分略)
4. Attention UNet
4.1 注意力机制背景
注意力机制能够自适应地聚焦于图像中的重要区域。在分割任务中,它可以抑制无关背景,增强目标区域特征,提高边界精确度。
4.2 Attention Gate原理
Attention UNet的核心是Attention Gate(AG)模块。它接收两个输入:来自解码器的粗粒度特征(门控信号g)和来自编码器的细粒度特征(x)。AG通过一系列线性变换和Sigmoid函数,为x的每个空间位置生成一个0到1之间的注意力系数α,然后对x进行加权:x̂ = x · α,从而让网络更关注与当前解码任务相关的区域。
4.3 网络架构
Attention UNet在标准UNet的基础上,将跳跃连接替换为带有Attention Gate的连接:编码器特征先经过AG(以解码器对应层特征为门控信号)进行加权,再与上采样后的特征进行拼接。
4.4 PyTorch代码实现
(此处展示AttentionGate模块和AttentionUNet网络的关键实现代码略)
5. 3D UNet
5.1 三维医学图像处理需求
CT、MRI等医学影像本质上是三维体积数据。2D分割方法逐切片处理会忽略切片间上下文,可能导致分割结果在三维空间中不连续。3D UNet应运而生,专门用于处理此类体积数据。
5.2 网络架构
3D UNet将UNet的所有2D操作(卷积、池化、批归一化、上采样)对等地扩展到3D维度,架构与2D UNet完全对应。
5.3 PyTorch代码实现
(此处展示3D版本的DoubleConv3D、Down3D、Up3D模块及UNet3D网络的关键实现代码略)
5.4 计算资源考量
3D UNet的计算和内存需求显著高于2D UNet。优化策略包括:减少基础通道数、使用小块(Patch)训练、采用混合精度训练以及梯度检查点技术。
6. 实验对比
6.1 常用数据集与评估指标
常用数据集包括ISBI 2012(神经元)、LiTS(肝脏肿瘤)、BraTS(脑肿瘤)等。主要评估指标有:
- Dice系数/F1分数:衡量重叠度。
- 交并比(IoU):另一种重叠度衡量标准。
- 豪斯多夫距离(HD):衡量分割边界的最大距离误差。
6.2 性能对比
综合多篇论文及复现实验,各变体典型性能对比如下(数据仅供参考):
- ISBI 2012:UNet++在Dice和IoU上通常略优于基础UNet和Attention UNet。
- LiTS肝脏分割:对于3D的CT数据,3D UNet在肝脏和肿瘤分割上的Dice分数均明显优于2D变体。
- BraTS脑肿瘤分割:3D UNet及其注意力改进版本在脑肿瘤各子区域的分割精度上超越2D UNet。
7. 应用场景
7.1 医学影像分割
这是UNet系列网络最初和最主要的应用领域,涵盖:
- CT图像分析:肝脏/肺部分割、骨组织分析。
- MRI图像分析:脑肿瘤分割、心脏结构分割。
- 病理图像分析:细胞核分割、血管网络提取。
在医疗AI领域,精准的分割结果是疾病诊断、手术规划和疗效评估的关键前提。
7.2 其他领域
- 卫星遥感:土地利用分类、建筑物与道路提取。
- 自动驾驶:可行驶区域、车道线及障碍物分割。
- 工业检测:产品表面缺陷检测、零件定位。
8. 总结与展望
8.1 各变体对比与选型建议
- UNet:通用性强,参数量适中,是优秀的基准模型和起点。
- UNet++:通过密集连接追求更精细的边界分割,参数量和计算量有所增加。
- Attention UNet:适用于需要精准目标定位的任务,能自适应聚焦关键区域。
- 3D UNet:专为三维体积数据设计,计算资源消耗大,但在处理CT/MRI等数据时必不可少。
选型流程参考:首先判断数据是否为3D体积数据(是则选3D UNet);对于2D数据,若任务强调精细边界可选UNet++,若强调目标定位可选Attention UNet,若追求效率与通用性则基础UNet仍是可靠选择。
8.2 未来发展方向
- 与Transformer融合:如TransUNet、Swin-UNet等,结合CNN的局部特征提取与Transformer的全局建模能力。
- 轻量化设计:面向移动端或边缘设备部署,如Mobile-UNet、通过知识蒸馏压缩模型。
- 减少标注依赖:探索自监督、半监督学习方法,以利用大量无标注数据。
9. 参考文献
- Ronneberger, O., Fischer, P., & Brox, T. (2015). U-Net: Convolutional Networks for Biomedical Image Segmentation. MICCAI.
- Zhou, Z., et al. (2018). UNet++: A Nested U-Net Architecture for Medical Image Segmentation. DLMIA.
- Oktay, O., et al. (2018). Attention U-Net: Learning Where to Look for the Pancreas. MIDL.
- Çiçek, Ö., et al. (2016). 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation. MICCAI.
- Chen, J., et al. (2021). TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation. arXiv.